Optimierung der 4C-seq-Datenanalyse: IMF fördert Bioinformatik-Projekt
Die Arbeitsgruppe Biomedical Informatics des Instituts für Medizinische Informatik (IMI) kann ihre erfolgreiche Forschung im Bereich der Analyse von räumlichen Chromosomen-Strukturen (“Circularized Chromosome Conformation Capture with sequencing”, 4C-seq) fortsetzen: Das Programm “Innovative Medizinische Forschung” des Universitätsklinikums Münster (UKM) fördert den IMI-Projektantrag zur Optimierung von 4C-seq-Datenanalysen für drei Jahre.
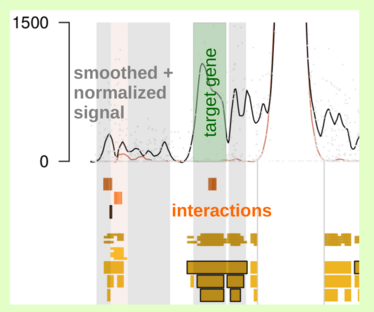
Die 4C-seq-Technologie gehört zu den aktuellen Hochdurchsatzmethoden („Next Generation Sequencing“, NGS), die im Zusammenspiel zwischen Medizin, Biologie und Informatik revolutionäre Einblicke in die Entstehung und Entwicklung von Krankheiten wie z.B. Krebs erlauben. Der epigenetische Datentyp 4C-seq ermöglicht es konkret, dreidimensionale Kontakte zwischen einem Punkt im Genom und dem Rest des Genoms zu untersuchen. Im Gegensatz zu genetischen Varianten sind diese epigenetischen Veränderungen potentiell umkehrbar, eine optimale Auswertung mittels Methoden der Informatik und Mathematik ist folglich auch für die Erforschung von neuen Behandlungsmethoden relevant. Die technische Auswertung von 4C-seq-Daten ist jedoch nicht einfach, es müssen verschiedene Eigenschaften der Daten berücksichtigt und bestimmte Fehlerquellen ausgeschlossen werden. Vorarbeiten aus einer langjährigen und erfolgreichen Kooperation des IMI mit dem Institut für Molekulare Tumorbiologie (IMTB) der Uniklinik Münster haben gezeigt, dass bereits existierende Programme noch nicht immer gute Ergebnisse für die Auswertung von 4C-seq-Daten liefern können. Das neu geförderte 4C-seq-Projekt hat daher zum Ziel, die technische Analyse von 4C-seq-Daten zu optimieren. Ein Schwerpunkt des Projekts ist die Erstellung und Optimierung eines neuen Algorithmus auf Grundlage bestehender Techniken, um die Analyse von 4C-seq-Daten zu verbessern – es sollen idealerweise weder richtige Interaktionen übersehen, noch zu viele falsche Interaktionen identifiziert werden. Weiterhin sollen neue Visualisierungsstrategien entwickelt werden, um einen guten Überblick über die gefundenen Regionen zu bieten. Das fertige Programm wird den eigentlichen Algorithmus und Visualisierungsoptionen mit einer benutzerfreundlichen Oberfläche verbinden und so die Projektergebnisse für interessierte Anwender aus den Bereichen der Bioinformatik oder Biologie bzw. Medizin bereitstellen.