Rekonstruktion der klonalen Evolution
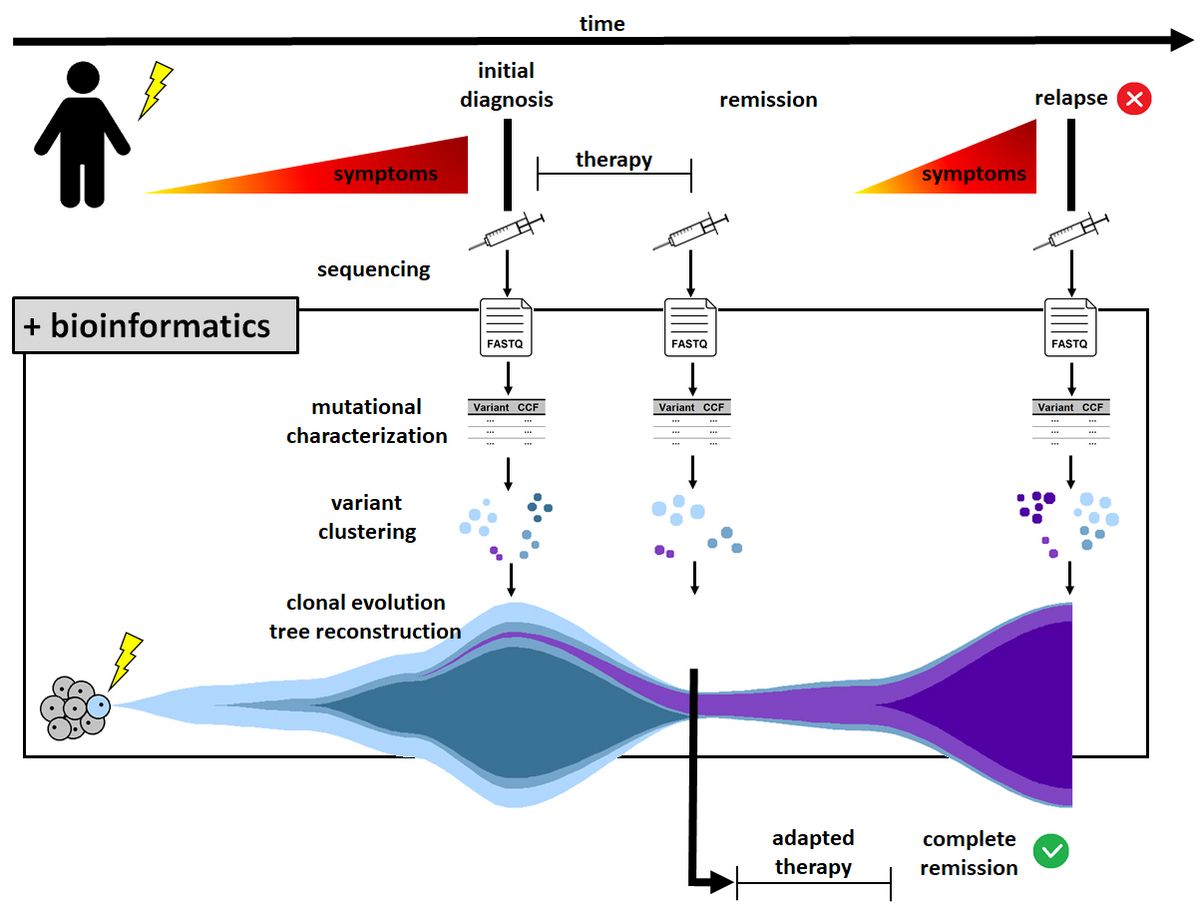
Im Rahmen der Krebsforschung ist die Analyse der klonalen Evolution eines Tumors von großem Interesse. Durch eine frühe Identifikation von aggressiven Subklonen können sich neuartige Therapieoptionen ergeben. Therapieresistenz kann unter Umständen bereits detektiert werden, bevor es zum Rückfall kommt, was einen erheblichen Einfluss auf das Überleben haben kann. Jedoch ist die eigentliche Rekonstruktion der klonalen Evolution auf Basis von Mutationsdaten nicht trivial. Für die Bioinformatik stellt sie eine Herausforderung dar.
Aktuell haben wir Kooperationen in folgenden klinischen und theoretischen Forschungsprojekten:
Analyse der klonalen Evolution bei adulten und pädiatrischen T-Lymphoblastischen Lymphomen, sowie bei adulter und pädiatrischer T-Zell akuter lymphatischer Leukämie; Kooperation mit der Klinik für Kinder- und Jugendmedizin - Pädiatrische Hämatologie und Onkologie des UKM (Univ.-Prof. Birgit Burkhardt) und dem Hämatologie Labor Kiel (Prof. Monika Brüggemann)
Analyse der klonalen Evolution bei pädiatrischem Myelodysplastischen Syndrom und akuter myeloischer Leukämie; Kooperation mit der Medizinischen Hochschule Hannover (Dr. Yvonne Behrens)
Optimierung der Algorithmen zur automatischen Rekonstruktion der klonalen Evolution; Kooperation mit dem Institut für Informatik der WWU (Univ.-Prof. Xiaoyi Jiang)
Analyse der klonalen Expansion in der männlichen Keimbahn; Kooperation mit der Clinical Research Unit „Male Germ Cells: From Genes to Function“ (Univ.-Prof. Schlatt und PD Dr. Nina Neuhaus)
Ergebnisse unserer Arbeit zur Analyse der klonalen Evolution wurden veröffentlicht:
Visualisierung der klonalen Evolution + automatische Schätzung von zusätzlichen Zeitpunkten und Therapieeffekten mit dem Programm „clevRvis“ (https://academic.oup.com/gigascience/article/doi/10.1093/gigascience/giad020/7113330, https://www.bioconductor.org/packages/clevRvis)
Systematische Evaluation von Programmen zur automatischen Analyse der klonalen Evolution basierend auf simulierten Daten + Simulations-Programm „clevRsim“ (https://www.mdpi.com/1660-4601/20/6/5128/html, https://github.com/sandmanns/clevRsim)
Klonale Evolution bei multiplem Myelom (https://www.frontiersin.org/articles/10.3389/fonc.2022.919278/full)
Klonale Evolution bei pädiatrischem Myelodysplastischen Syndrom und akuter myeloischer Leukämie (https://www.frontiersin.org/articles/10.3389/fonc.2022.888114/full)
Evaluation von Programmen zur automatischen Analyse der klonalen Evolution basierend auf exemplarischen echten Daten (https://cgp.iiarjournals.org/content/19/2/194.long)
Klonale Evolution beim Burkitt-Lymphom (https://www.nature.com/articles/s41375-020-0862-5)
Klonale Evolution bei adultem Myelodysplastischen Syndrom (https://www.nature.com/articles/ncomms15099)
Kontakt: PD Dr. Sarah Sandmann


