News
Auf der Suche nach dem Ur-Sprung: Münstersches Projekt zur Genomanalyse soll mehr Licht ins Dunkel der Evolution bringen
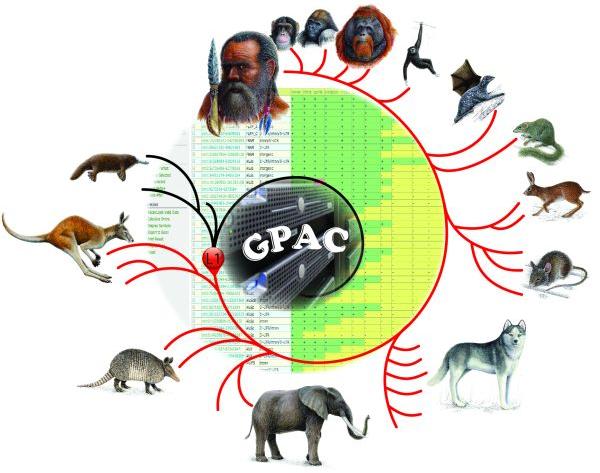
Münster (mfm/ps) - Springende Gene: Was sich wie die biologische Ausstattung von Kängurus anhört, steckt in jedem Säugetier - und macht auch mehr als die Hälfte unseres Erbgutes aus. Die Forschergruppe um den Molekularbiologen PD Dr. Jürgen Schmitz von der Medizinischen Fakultät der Universität Münster nutzt diese Gene, um die bereits viele Millionen Jahre währende Entwicklung vom Ursäuger bis zum Menschen zu erhellen. „Die Geschichte der Moleküle rekonstruieren, um die Phylogenese, also die Stammesgeschichte der Arten, zu vervollständigen“ – das ist laut Schmitz der Leitsatz seines neuen Projektes, das von der Deutschen Forschungsgemeinschaft (DFG) eine großzügige Finanzspritze von gut 463.000 Euro bekommt.
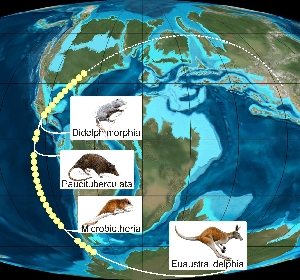
Wo die springenden Gene, fachlich Retroposons genannt, ursprünglich herkommen, weiß niemand genau. Es wird vermutet, dass es sich um domestizierte Viren handelt, die in der Zelle geblieben sind. Sie haben irgendwann begonnen, sich erfolgreich selbst zu kopieren und sich an beliebigen Stellen des DNA-Moleküls „dazwischen zu drängeln“. Retroposons werden auch als „Fossile im Genom“ bezeichnet: Sie sind oft über viele Millionen Jahre aktiv und können über die Keimbahn vererbt werden, weswegen man mit ihnen die Verwandtschaft verschiedener Arten sehr zielsicher rekonstruieren kann. Dies wiederum ist die Grundvoraussetzung, um sinnvolle vergleichende Studien in vielen Bereichen der Wissenschaft einschließlich der Medizin durchführen zu können.
Hierfür werden die Genome der betreffenden Organismen aneinandergelegt (aligniert) und auf gemeinsame Retroposons untersucht, die sich an derselben Stelle im Genom befinden und von einem gemeinsamen Vorfahren abstammen. So konnte zum Beispiel erstmals sicher gezeigt werden, dass Mensch und Schimpanse einen gemeinsamen Vorfahren haben, zugleich beide aber nur entfernt mit dem Gorilla verwandt sind. Durch Analyse und Vergleich dieser informativen Retropsons sind die Wissenschaftler in der Lage, den Zeitpunkt des Gensprunges zu ermitteln und so vergleichend die Phylogenese zum Menschen und anderer Arten zu entschlüsseln.
Das Fördergeld der DFG soll dazu verwendet werden, einen internetbasierten Retrogenom-Server und Datenbanken zur automatisierten Klärung evolutiver Fragen zu schaffen. Forscher können DNA-Sequenzen oder schlicht den Namen eines Organismus online in das System einspeisen und bekommen Informationen über enthaltene Retroposons. „Des Weiteren wird den Nutzern angezeigt, wo sich der betreffende Organismus im Artenstammbaum gezielt einordnen lässt“, schildert PD Jürgen Schmitz die Möglichkeiten des bis 2019 geförderten Projektes. Zusammen mit Kollegen aus dem Institut für Bioinformatik der Universität Münster arbeitet die Forschergruppe an der komplexen Internet-Plattform, die bestehende Analyse- und Datenbankmodule mit neuen Elementen kombiniert und der wissenschaftlichen Gemeinschaft dann frei zur Verfügung steht.
Nicht nur Evolutionsforscher, sondern auch Mediziner können von diesem Projekt profitieren: Da sich die springenden Gene zuweilen auch in lebenswichtigen Gensequenzen einnisten, sind sie auch Ursache von Erbkrankheiten. Mit Hilfe der Retroposon-Analyse können die Forscher herausfinden, wann die Krankheit auf der Linie zum Menschen ihren Ursprung und wie sich die ausschlaggebende Änderung von Primatenlinie zu Primatenlinie bis zum eigentlichen Krankheitsbild im Menschen entwickelt hat.
Wo die springenden Gene, fachlich Retroposons genannt, ursprünglich herkommen, weiß niemand genau. Es wird vermutet, dass es sich um domestizierte Viren handelt, die in der Zelle geblieben sind. Sie haben irgendwann begonnen, sich erfolgreich selbst zu kopieren und sich an beliebigen Stellen des DNA-Moleküls „dazwischen zu drängeln“. Retroposons werden auch als „Fossile im Genom“ bezeichnet: Sie sind oft über viele Millionen Jahre aktiv und können über die Keimbahn vererbt werden, weswegen man mit ihnen die Verwandtschaft verschiedener Arten sehr zielsicher rekonstruieren kann. Dies wiederum ist die Grundvoraussetzung, um sinnvolle vergleichende Studien in vielen Bereichen der Wissenschaft einschließlich der Medizin durchführen zu können.
Hierfür werden die Genome der betreffenden Organismen aneinandergelegt (aligniert) und auf gemeinsame Retroposons untersucht, die sich an derselben Stelle im Genom befinden und von einem gemeinsamen Vorfahren abstammen. So konnte zum Beispiel erstmals sicher gezeigt werden, dass Mensch und Schimpanse einen gemeinsamen Vorfahren haben, zugleich beide aber nur entfernt mit dem Gorilla verwandt sind. Durch Analyse und Vergleich dieser informativen Retropsons sind die Wissenschaftler in der Lage, den Zeitpunkt des Gensprunges zu ermitteln und so vergleichend die Phylogenese zum Menschen und anderer Arten zu entschlüsseln.
Das Fördergeld der DFG soll dazu verwendet werden, einen internetbasierten Retrogenom-Server und Datenbanken zur automatisierten Klärung evolutiver Fragen zu schaffen. Forscher können DNA-Sequenzen oder schlicht den Namen eines Organismus online in das System einspeisen und bekommen Informationen über enthaltene Retroposons. „Des Weiteren wird den Nutzern angezeigt, wo sich der betreffende Organismus im Artenstammbaum gezielt einordnen lässt“, schildert PD Jürgen Schmitz die Möglichkeiten des bis 2019 geförderten Projektes. Zusammen mit Kollegen aus dem Institut für Bioinformatik der Universität Münster arbeitet die Forschergruppe an der komplexen Internet-Plattform, die bestehende Analyse- und Datenbankmodule mit neuen Elementen kombiniert und der wissenschaftlichen Gemeinschaft dann frei zur Verfügung steht.
Nicht nur Evolutionsforscher, sondern auch Mediziner können von diesem Projekt profitieren: Da sich die springenden Gene zuweilen auch in lebenswichtigen Gensequenzen einnisten, sind sie auch Ursache von Erbkrankheiten. Mit Hilfe der Retroposon-Analyse können die Forscher herausfinden, wann die Krankheit auf der Linie zum Menschen ihren Ursprung und wie sich die ausschlaggebende Änderung von Primatenlinie zu Primatenlinie bis zum eigentlichen Krankheitsbild im Menschen entwickelt hat.