Forschungsthematik
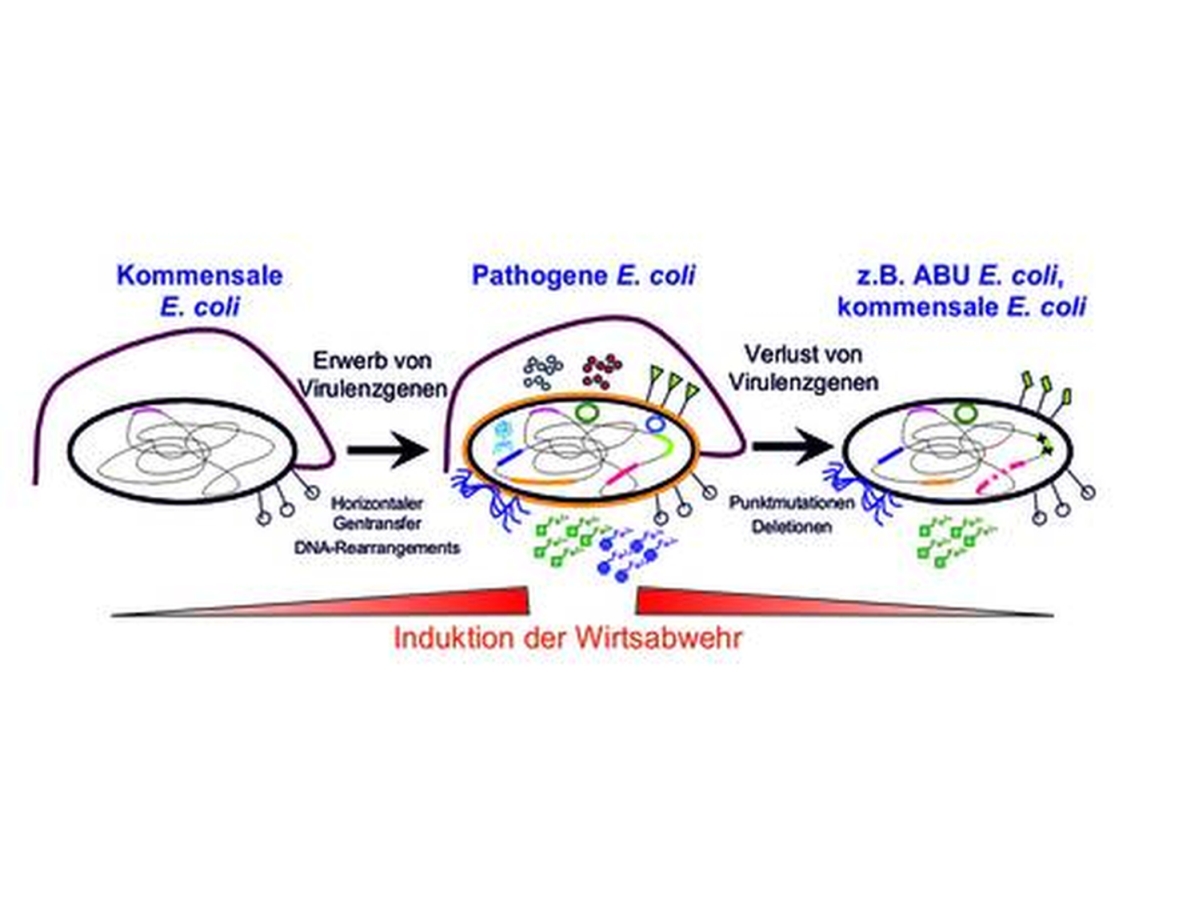
Was unterscheidet pathogene von kommensalen Bakterien? Welche Eigenschaften benötigen Bakterien, um symptomatische bzw. subklinische Infektionen auszulösen? Verändern sich Bakterien im Verlauf einer Infektion?
Um diese Fragestellungen zu beantworten, verwenden wir Escherichia coli als Modellorganismus, da diese Spezies nicht nur apathogene kommensale, sondern auch sogenannte extraintestinal pathogene E. coli (ExPEC) und intestinal pathogene E. coli (IPEC)-Varianten umfasst. Während eine IPEC-Infektion zu Durchfallerkrankungen führt, können ExPEC vor allem Harnwegsinfektionen (die häufigste bakterielle Infektion in industrialisierten Ländern) und Meningitis bei Neugeborenen verursachen. Darüber hinaus sind ExPEC auch häufig als Erreger nosokomialer Infektionen oder Septikämien von großer Bedeutung. Die ständig zunehmende Zahl und Verbreitung (multi-)resistenter Varianten stellt ein großes Problem bei der Behandlung von E. coli-Infektionen dar.
Wir charakterisieren mit Hilfe funktionaler und vergleichender Genomanalysen Virulenz- und Fitness-assoziierte Gene bei ExPEC sowie ausgewählten intestinalen E. coli Pathotypen und bearbeiten grundlegende Mechanismen der Bakterien-Wirt-Interaktion, die an der Kolonisierung bzw. an der Entstehung symptomatischer oder chronischer E. coli-Infektionen beteiligt sind.
In diesem Zusammenhang interessieren wir uns für Unterschiede im Genomgehalt und der Genomstruktur, welche neue Ansätze für eine verbesserte Diagnostik, Risikobewertung oder Therapie bzw. Prävention von ExPEC Infektionen liefern können. Weiterhin untersuchen wir Genregulationsprozesse, metabolische Eigenschaften und Interaktionsstrategien pathogener E. coli mit Eukaryonten, welche kommensale Varianten von Pathogenen bzw. unterschiedliche Pathotypen voneinander unterscheiden.
Ein weiterer Schwerpunkt liegt auf der Untersuchung der mikrobiellen Genomplastizität und ihrer Bedeutung für (i) die Entstehung und Ausprägung von Antibiotika-Resistenzen, (ii) die mikrobielle Pathogenität und (iii) die Adaptation (Mikroevolution) an unterschiedliche Nischen und Lebensräume.
Um diese Fragestellungen zu beantworten, verwenden wir Escherichia coli als Modellorganismus, da diese Spezies nicht nur apathogene kommensale, sondern auch sogenannte extraintestinal pathogene E. coli (ExPEC) und intestinal pathogene E. coli (IPEC)-Varianten umfasst. Während eine IPEC-Infektion zu Durchfallerkrankungen führt, können ExPEC vor allem Harnwegsinfektionen (die häufigste bakterielle Infektion in industrialisierten Ländern) und Meningitis bei Neugeborenen verursachen. Darüber hinaus sind ExPEC auch häufig als Erreger nosokomialer Infektionen oder Septikämien von großer Bedeutung. Die ständig zunehmende Zahl und Verbreitung (multi-)resistenter Varianten stellt ein großes Problem bei der Behandlung von E. coli-Infektionen dar.
Wir charakterisieren mit Hilfe funktionaler und vergleichender Genomanalysen Virulenz- und Fitness-assoziierte Gene bei ExPEC sowie ausgewählten intestinalen E. coli Pathotypen und bearbeiten grundlegende Mechanismen der Bakterien-Wirt-Interaktion, die an der Kolonisierung bzw. an der Entstehung symptomatischer oder chronischer E. coli-Infektionen beteiligt sind.
In diesem Zusammenhang interessieren wir uns für Unterschiede im Genomgehalt und der Genomstruktur, welche neue Ansätze für eine verbesserte Diagnostik, Risikobewertung oder Therapie bzw. Prävention von ExPEC Infektionen liefern können. Weiterhin untersuchen wir Genregulationsprozesse, metabolische Eigenschaften und Interaktionsstrategien pathogener E. coli mit Eukaryonten, welche kommensale Varianten von Pathogenen bzw. unterschiedliche Pathotypen voneinander unterscheiden.
Ein weiterer Schwerpunkt liegt auf der Untersuchung der mikrobiellen Genomplastizität und ihrer Bedeutung für (i) die Entstehung und Ausprägung von Antibiotika-Resistenzen, (ii) die mikrobielle Pathogenität und (iii) die Adaptation (Mikroevolution) an unterschiedliche Nischen und Lebensräume.