News
„Protein-Fingerabdruck“ des Meisters der Regeneration: MPI gelingt weiterer Erfolg in der Stammzellforschung
Münster (mpi) - Planarien sind Plattwürmer, die seit langem als Meister der Regeneration bekannt sind: Nach einer Amputation können sie quasi jedes Körperteil oder Organ neu bilden. Ihre Fähigkeit zur Regeneration basiert auf einer großen Menge an adulten pluripotenten Stammzellen, zellulären Alleskönnern, die fast überall im Tier vorkommen. Dadurch eignen sich Planarien hervorragend als Modellorganismen in der Stammzellforschung. Allerdings ist der Plattwurm als Modellorganismus methodisch bislang nur unzureichend etabliert. Dr. Kerstin Bartscherer, Leiterin einer Forschungsgruppe am Max-Planck-Institut (MPI) für molekulare Biomedizin in Münster, und Kollegen haben deshalb eine Methode zur Analyse von „Protein-Fingerabdrücken“ in Planarien entwickelt. Die Forscher entdeckten dabei ein Protein, das nicht nur ausreichenden Stammzellvorrat in Planarien gewährleistet, sondern auch im Zellkern von pluripotenten Stammzellen in Mausembryonen vorkommt (Cell Reports, online 21.11.2013).
„Bei Planarien können wir pluripotente Stammzellen im adulten Organismus manipulieren – das ist mit Säugetieren nicht möglich: Bei ihnen können pluripotente Stammzellen nur im frühen Embryo oder in der Kulturschale untersucht werden“, lobt Dr. Bartscherer die Vorzüge der Plattwürmer. Um aber zu wissen, welche Proteine überhaupt für die pluripotenten Eigenschaften ihrer Stammzellen und damit für die außergewöhnlichen Regenerationsfähigkeiten der Planarien eine Rolle spielen könnten, müssen diese Proteine von Grund auf untersucht werden. Das Team um Bartscherer musste also das „Stammzellproteom“, eine Art „Fingerabdruck“ von Stammzellproteinen, bestimmen und die für die Stammzellerhaltung relevanten Proteine identifizieren. Es hat dazu eine effiziente Art der Proteinmarkierung, die SILAC-Methode, zusammen mit hochauflösender Massenspektrometrie eingesetzt, mit deren Hilfe innerhalb weniger Wochen Zehntausende von Proteinfragmenten anhand ihrer Masse identifiziert und den jeweiligen Proteinen zugeordnet werden können (SILAC steht für Stable Isotope Labeling with Aminoacids in Culture).
„Die quantitative Massenspektrometrie der Proteine von unbekannteren Modellorganismen wie dem Plattwurm ist eine gute Methode, um unvoreingenommen biologische Prozesse zu beleuchten und die daran beteiligten Proteine zu identifizieren. Viele Methoden und Werkzeuge, die für andere Organismen selbstverständlich sind, stehen derzeit für diesen Organismus einfach nicht zur Verfügung,“ erklärt Dr. Hannes Drexler, Leiter der Proteomics-Abteilung am MPI, den gezielten Einsatz der SILAC-Methode.
Alexander Böser, Doktorand und Erstautor der Studie, erklärt die genaue Vorgehensweise: „Zunächst mussten alle Proteine in den lebenden Tieren über den Stoffwechsel mit einer ‚schweren’ Aminosäure markiert werden. Dies gelang uns durch die Fütterung mit Leber von speziellen Mäusen, den SILAC-Mäusen, deren Proteine bereits vollständig mit der ‚schweren’ Aminosäure markiert waren.“
Um besonders die Proteine zu finden, die angereichert in den pluripotenten Stammzellen der Plattwürmer vorkommen, wurde mit der neu entwickelten Methode die Häufigkeit der Proteine in Stammzellen und die in bereits spezialisierten Zellen gemessen und miteinander verglichen. Bartscherer freut sich über das Ergebnis: „Durch diesen Vergleich von markierten und nicht markierten Plattwurmextrakten haben wir fast 400 mögliche Stammzellproteine gefunden, von denen wir einen Teil wiederum durch andere experimentelle Ansätze bestätigt haben.“ Tatsächlich wurden mehrere Proteine aus diesem Datensatz zum ersten Mal mit Stammzellen in Verbindung gebracht. Unter diesen waren auch einige Proteine, die in ähnlicher Form in Säugetieren vorkommen.
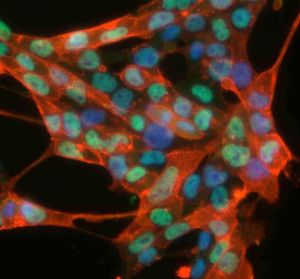
Eines dieser Proteine nahmen die Wissenschaftler genauer unter die Lupe: das Zellkernprotein Nuclear Receptor Co-activator 5, kurz „Ncoa5“. Unter den Zellkernproteinen gibt es besonders „mächtige“ Proteine, welche die „Befehle“, die eine Zelle aus der extrazellulären Umgebung erhält, durch die Regulation von Genexpression ausführen können. Eine Veränderung der Genexpression kann das Verhalten einer Zelle stark beeinflussen und zum Beispiel aus einer pluripotenten, also alleskönnenden Stammzelle, eine spezialisierte Nerven- oder Muskelzelle machen.
„Durch das gezielte Ausschalten von Ncoa5 in Planarien konnten wir zeigen, dass Ncoa5 für die Erhaltung des pluripotenten Stammzellvorrates wichtig ist. Planarien, die kein Ncoa5 Protein produzieren konnten, büßten sämtliche Stammzellen über einen Zeitraum von wenigen Wochen ein und konnten kaum Gewebe regenerieren“, sagt Böser. In Zusammenarbeit mit Wissenschaftlern um Luca Gentile und Hans Schöler - ebenfalls am MPI Münster - konnte interessanterweise durch Experimente mit Mausembryonen Ncoa5 erstmals auch im Säugetier den Stammzellen zugeordnet werden. „Unsere Ergebnisse zeigen, dass die Forschung mit Plattwürmern durchaus relevante Ergebnisse zur allgemeinen Stammzellforschung beitragen kann“, so Bartscherer.
Durch welchen Mechanismus Ncoa5 zur Erhaltung des Stammzellpools in Planarien beiträgt, wird derzeit erforscht. “Besonders spannend wird es sein, die Frage zu beantworten, ob Ncoa5 auch in Säugetieren für die Pluripotenz und somit zur Erhaltung der embryonalen Stammzellen benötigt wird“, freut sich Kerstin Bartscherer auf zukünftige Studien.
Link zur Publikation
„Bei Planarien können wir pluripotente Stammzellen im adulten Organismus manipulieren – das ist mit Säugetieren nicht möglich: Bei ihnen können pluripotente Stammzellen nur im frühen Embryo oder in der Kulturschale untersucht werden“, lobt Dr. Bartscherer die Vorzüge der Plattwürmer. Um aber zu wissen, welche Proteine überhaupt für die pluripotenten Eigenschaften ihrer Stammzellen und damit für die außergewöhnlichen Regenerationsfähigkeiten der Planarien eine Rolle spielen könnten, müssen diese Proteine von Grund auf untersucht werden. Das Team um Bartscherer musste also das „Stammzellproteom“, eine Art „Fingerabdruck“ von Stammzellproteinen, bestimmen und die für die Stammzellerhaltung relevanten Proteine identifizieren. Es hat dazu eine effiziente Art der Proteinmarkierung, die SILAC-Methode, zusammen mit hochauflösender Massenspektrometrie eingesetzt, mit deren Hilfe innerhalb weniger Wochen Zehntausende von Proteinfragmenten anhand ihrer Masse identifiziert und den jeweiligen Proteinen zugeordnet werden können (SILAC steht für Stable Isotope Labeling with Aminoacids in Culture).
„Die quantitative Massenspektrometrie der Proteine von unbekannteren Modellorganismen wie dem Plattwurm ist eine gute Methode, um unvoreingenommen biologische Prozesse zu beleuchten und die daran beteiligten Proteine zu identifizieren. Viele Methoden und Werkzeuge, die für andere Organismen selbstverständlich sind, stehen derzeit für diesen Organismus einfach nicht zur Verfügung,“ erklärt Dr. Hannes Drexler, Leiter der Proteomics-Abteilung am MPI, den gezielten Einsatz der SILAC-Methode.
Alexander Böser, Doktorand und Erstautor der Studie, erklärt die genaue Vorgehensweise: „Zunächst mussten alle Proteine in den lebenden Tieren über den Stoffwechsel mit einer ‚schweren’ Aminosäure markiert werden. Dies gelang uns durch die Fütterung mit Leber von speziellen Mäusen, den SILAC-Mäusen, deren Proteine bereits vollständig mit der ‚schweren’ Aminosäure markiert waren.“
Um besonders die Proteine zu finden, die angereichert in den pluripotenten Stammzellen der Plattwürmer vorkommen, wurde mit der neu entwickelten Methode die Häufigkeit der Proteine in Stammzellen und die in bereits spezialisierten Zellen gemessen und miteinander verglichen. Bartscherer freut sich über das Ergebnis: „Durch diesen Vergleich von markierten und nicht markierten Plattwurmextrakten haben wir fast 400 mögliche Stammzellproteine gefunden, von denen wir einen Teil wiederum durch andere experimentelle Ansätze bestätigt haben.“ Tatsächlich wurden mehrere Proteine aus diesem Datensatz zum ersten Mal mit Stammzellen in Verbindung gebracht. Unter diesen waren auch einige Proteine, die in ähnlicher Form in Säugetieren vorkommen.
Eines dieser Proteine nahmen die Wissenschaftler genauer unter die Lupe: das Zellkernprotein Nuclear Receptor Co-activator 5, kurz „Ncoa5“. Unter den Zellkernproteinen gibt es besonders „mächtige“ Proteine, welche die „Befehle“, die eine Zelle aus der extrazellulären Umgebung erhält, durch die Regulation von Genexpression ausführen können. Eine Veränderung der Genexpression kann das Verhalten einer Zelle stark beeinflussen und zum Beispiel aus einer pluripotenten, also alleskönnenden Stammzelle, eine spezialisierte Nerven- oder Muskelzelle machen.
„Durch das gezielte Ausschalten von Ncoa5 in Planarien konnten wir zeigen, dass Ncoa5 für die Erhaltung des pluripotenten Stammzellvorrates wichtig ist. Planarien, die kein Ncoa5 Protein produzieren konnten, büßten sämtliche Stammzellen über einen Zeitraum von wenigen Wochen ein und konnten kaum Gewebe regenerieren“, sagt Böser. In Zusammenarbeit mit Wissenschaftlern um Luca Gentile und Hans Schöler - ebenfalls am MPI Münster - konnte interessanterweise durch Experimente mit Mausembryonen Ncoa5 erstmals auch im Säugetier den Stammzellen zugeordnet werden. „Unsere Ergebnisse zeigen, dass die Forschung mit Plattwürmern durchaus relevante Ergebnisse zur allgemeinen Stammzellforschung beitragen kann“, so Bartscherer.
Durch welchen Mechanismus Ncoa5 zur Erhaltung des Stammzellpools in Planarien beiträgt, wird derzeit erforscht. “Besonders spannend wird es sein, die Frage zu beantworten, ob Ncoa5 auch in Säugetieren für die Pluripotenz und somit zur Erhaltung der embryonalen Stammzellen benötigt wird“, freut sich Kerstin Bartscherer auf zukünftige Studien.
Link zur Publikation